Octubre, 2024
La Real Academia Sueca de las Ciencias ha concedido en Nobel de Química 2024 al bioquímico estadounidense David Baker por diseñar desde cero nuevas proteínas mediante su programa RoseTTAFold. La otra mitad la comparten el británico Demis Hassabis y el también estadounidense John M. Jumper, ambos de la empresa DeepMind de Google, que lograron predecir las estructura proteicas con IA.
Este año el Nobel de Química se ha centrado en las proteínas, los componentes básicos de la vida.
Estos compuestos se encuentran en todas las células del cuerpo humano y controlan y dirigen todas las reacciones químicas que, en conjunto, constituyen la base de nuestra existencia.
El Comité del Nobel las describe, con justa razón, como “las ingeniosas herramientas químicas de la vida”.
Y sí, los son.
Por ello, ha decidido conceder la mitad del Nobel de Química 2024 al bioquímico estadounidense David Baker (Seattle, Washington, 1962), director del Institute for Protein Design de la Universidad de Washington.
Baker es el creador de RoseTTAFold, lanzado en 2021 por su laboratorio. Este programa es capaz no sólo de desvelar la compleja estructura 3D que adopta una cadena de aminoácidos al plegarse para dar lugar a una proteína funcional, sino también de diseñar otras completamente nuevas desde cero. Este avance, decía en una entrevista de 2023, “tiene un gran potencial en campos como el medioambiente, la energía y, sobre todo, en biomedicina”.
La otra mitad del galardón la comparten de forma conjunta Demis Hassabis (Londres, 1976) y John Michael Jumper (Arkansas, 1985) investigadores de la empresa británica DeepMind, propiedad de Google. El jurado ha distinguido a ambos por sus contribuciones al uso de inteligencia artificial (IA) de aprendizaje profundo para la predicción exacta de la estructura tridimensional de las proteínas.
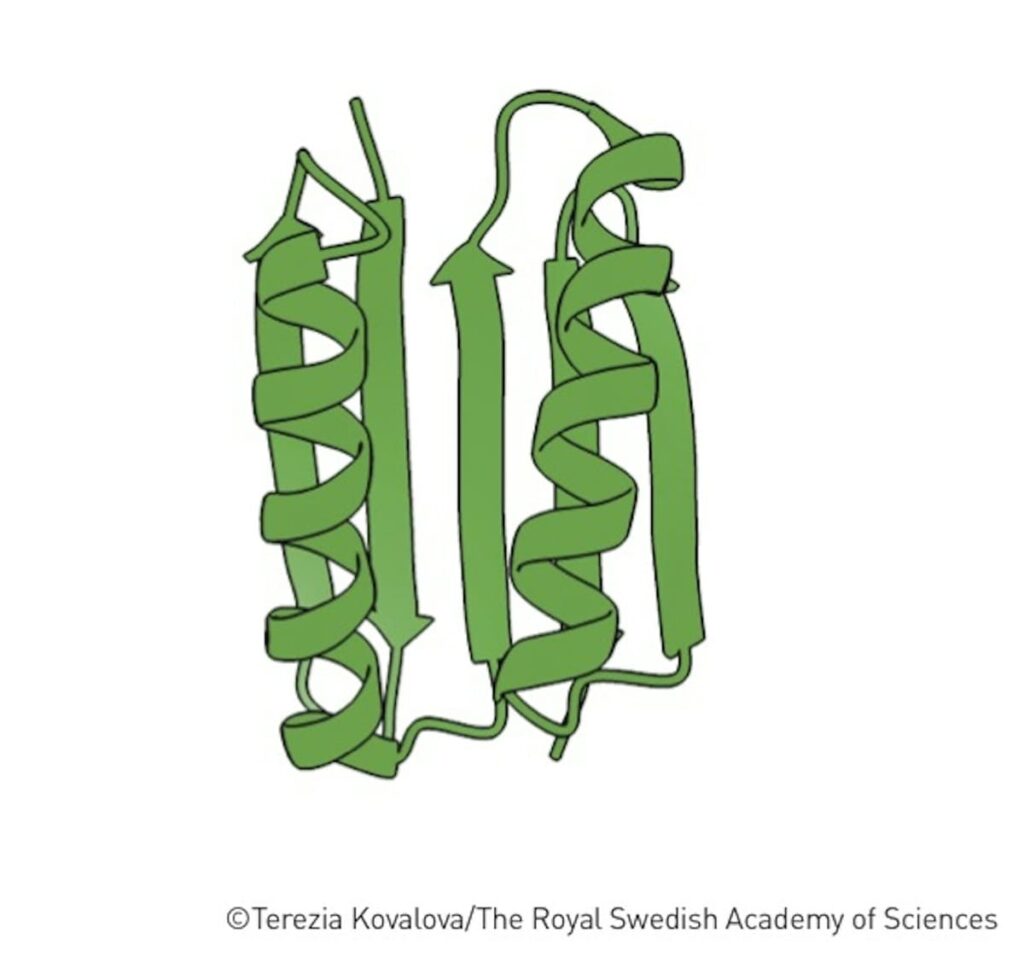
Proteínas sintéticas con múltiples aplicaciones
Heiner Linke, presidente del Comité Nobel de Química, ha sido claro: “Uno de los descubrimientos reconocidos este año se refiere a la construcción de proteínas espectaculares. El otro consiste en hacer realidad un sueño de 50 años: predecir las estructuras de las proteínas a partir de sus secuencias de aminoácidos. Ambos descubrimientos abren enormes posibilidades”.
Las proteínas suelen estar formadas por 20 aminoácidos diferentes, que pueden describirse como los componentes básicos de la vida. En 2003, Baker consiguió utilizar estos bloques para diseñar una nueva proteína que no se parecía a ninguna otra. Desde entonces, su grupo de investigación ha producido una creación proteínica imaginativa tras otra, incluidas proteínas que pueden utilizarse como fármacos, vacunas, nanomateriales y sensores diminutos, mediante RoseTTAFold y nuevas versiones del programa.
El segundo descubrimiento se refiere a la predicción de estructuras proteicas. En las proteínas, los aminoácidos están unidos en largas cadenas que se pliegan para formar una estructura tridimensional, decisiva para la función de la proteína.
Desde los años setenta, los investigadores habían intentado predecir las estructuras de las proteínas a partir de secuencias de aminoácidos, pero resultaba notoriamente difícil. Sin embargo, hace cuatro años se produjo un avance asombroso.
En 2020, Hassabis y Jumper presentaron un modelo de IA llamado AlphaFold2. Con su ayuda, han podido predecir la estructura de prácticamente todos los 200 millones de proteínas que los investigadores han identificado. Desde su descubrimiento, AlphaFold2 ha sido utilizado por más de dos millones de personas de 190 países. Entre un sinfín de aplicaciones científicas, los investigadores pueden ahora comprender mejor la resistencia a los antibióticos y crear imágenes de enzimas capaces de descomponer el plástico.
En su comunicado, la Academia Sueca ha puntualizado: “La vida no podría existir sin proteínas. Que ahora podamos predecir sus estructuras y diseñar nuestras propias proteínas confiere el mayor beneficio a la humanidad”. (Redacción SdE / Agencia SINC)
▪️◾▪️
Nobel de Química a la estructura de las proteínas una vez más… ¿la última?
Antonio M. Rodríguez García / Antonio de la Hoz Ayuso / Enrique Díez Barra
Las proteínas, esas conocidas moléculas compuestas por secuencias de aminoácidos, están presentes en nuestra alimentación cotidiana y son muy importantes en todos los procesos de la vida. Las combinaciones de los 20 aminoácidos esenciales —desde decenas a miles— generan todas ellas.
El estudio de su composición, función y estructura recibe la atención de los investigadores y de la de Academia Sueca de Ciencias desde hace mucho tiempo: lo han reconocido en los premios Nobel de 1957, 1962, 1972, 1982, 1983, 2004, 2012 y, ahora, 2024.
¿Será este el último premio Nobel relacionado con las proteínas? Muy probablemente, ya que la contribución de David Baker, Demis Hassabis y John M. Jumper, los galardonados de este año, ha resuelto de un plumazo dos de los grandes retos de la bioquímica.

Por un lado, ha permitido conocer la estructura tridimensional de unas 200 000 proteínas —prácticamente, todas las conocidas— con ayuda de la inteligencia artificial. Por otro, hace posible “crear” proteínas, esto es, identificar la secuencia de aminoácidos que forma una proteína con la estructura tridimensional deseada.
Deconstruyendo los ladrillos de la vida
¿Y para qué interesa conocer la estructura tridimensional de las proteínas y tener la capacidad de crearlas a demanda? Para el beneficio de la humanidad, como reclamaba Alfred Nobel en el testamento que da origen a estos galardones.
Saber por qué se produce la resistencia a antibióticos, cuál es el mecanismo para que se desarrollen determinadas enfermedades, entender cómo algunas bacterias descomponen material plástico, acelerar los procedimientos para crear vacunas, crear fármacos específicos o personalizados o nuevos nanomateriales, es, tras las aportaciones de los galardonados, mucho más fácil, rápido y accesible al conjunto de los investigadores de diversos campos.
Para poder construir desde cero
David Baker es especialista en desarrollar programas de química computacional para, estableciendo la premisa de la estructura 3D deseada, identificar qué aminoácidos componen la estructura primaria de la proteína.
En las propias palabras de Baker: “Si quieres construir un avión, no empiezas modificando un pájaro, sino que comprendes los primeros principios de la aerodinámica y construyes máquinas voladoras a partir de ellos”. Esto cambió ya en 2003 la estrategia antes basada en la modificación de proteínas conocidas.
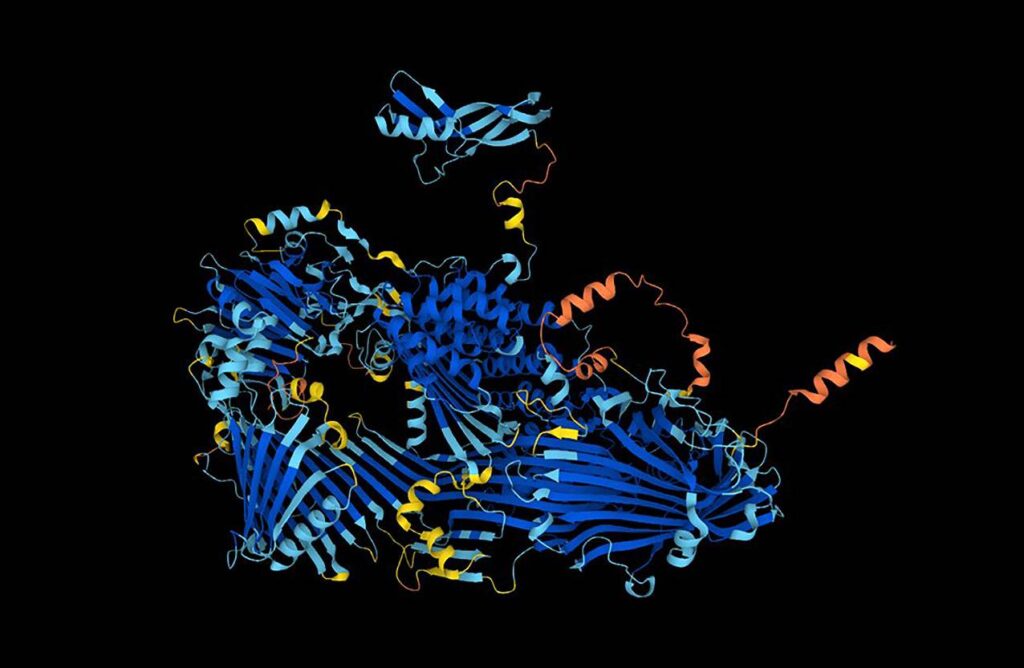
Por su parte Demis Hassabis y John Jumper, este con menos de 40 años, han aprovechado los desarrollos de la inteligencia artificial inicialmente aplicados en juegos de mesa.
El programa Deep Blue consiguió derrotar al campeón del mundo de ajedrez Gary Kasparov en 1997 y después se retiró, ya que no era ese su objetivo final. De la misma manera Alpha Go consiguió derrotar al campeón del mundo de GO en 2016. El primero utilizaba la potencia de cálculo y bases de datos de partidas. El segundo conseguía aprender a partir de las reglas del juego.
Con estas mismas herramientas, los científicos abordan el objetivo final: desentrañar la estructura de moléculas complejas como las proteínas y conseguir diseñar nuevas estructuras y aplicaciones de estas moléculas tan transcendentales para la vida.
El equipo Hassabis/Jumper entrenó al sistema de inteligencia artificial AlphaFold2 con la enorme información de las bases de datos de todas las estructuras de proteínas y secuencias de aminoácidos conocidas y la nueva arquitectura de IA empezó a dar buenos resultados. Esta versión mejorada logró un hito histórico en el CASP de 2020 —algo así como unas olimpiadas de proteínas—, alcanzando una precisión comparable a la cristalografía de rayos X y resolviendo efectivamente un desafío de 50 años en la predicción de estructuras proteicas. ![]()





